
Главная страница Случайная страница
Разделы сайта
АвтомобилиАстрономияБиологияГеографияДом и садДругие языкиДругоеИнформатикаИсторияКультураЛитератураЛогикаМатематикаМедицинаМеталлургияМеханикаОбразованиеОхрана трудаПедагогикаПолитикаПравоПсихологияРелигияРиторикаСоциологияСпортСтроительствоТехнологияТуризмФизикаФилософияФинансыХимияЧерчениеЭкологияЭкономикаЭлектроника
Свойства и функции ДНК-топоизмераз
|
|
| Организм | Фермент | Ген | Поло-жение гена* | Длина белка (а.о.) | Субъеди-ничная структура | Подсе-мейство | Функции |
| E. coli | Топоизомераза I | topA | мономер | IA | |||
| ДНК-гираза | gyrA | гетеро-тетрамер А2В2 | IIA | ||||
| gyrB | |||||||
| Топоизомераза III | topB | мономер | IA | ||||
| Топоизомераза IV | parC | гетеро- тетрамер С2Е2 | IIA | ||||
| parE | |||||||
| S. cerevisiae | Топоизомераза I | ТОР1 | XV | мономер | IA | ||
| Топоизомераза II | ТОР2 | XIV | гомодимер | IIA | |||
| Топоизомераза III | ТОР3 | XII | мономер | IA | |||
| Человек | Топоизомераза I | ТОР1 | мономер | IB | |||
| Топоизомераза IIa | ТОР2a | 17q21-22 | гомодимер | IIA | |||
| Топоизомераза IIb | ТОР2b | 3p24 | гомодимер | IIA | |||
| Топоизомераза IIIa | ТОР3a | 17p11-12 | мономер | IA | |||
| Топоизомераза IIIb | ТОР3b | мономер | IA |
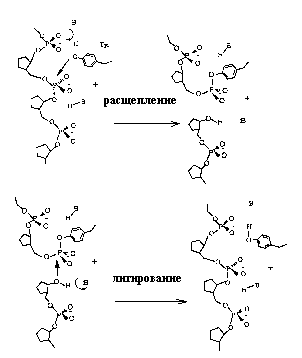
Рис. 2.25. Механизмы стадий расщепления и лигирования нити ДНК ДНК-топоизомеразами
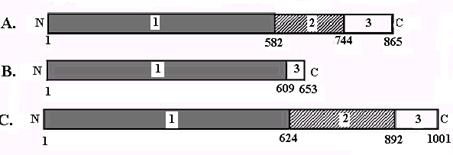
Рис. 2.26. Доменная структура ДНК-топоизомераз типа IA.
А – ДНК-топоизомераза I E. coli, B – ДНК-топоизомераза III E. coli, C – ДНК-топоизомераза IIIa человека.
1 – домен расщепления и прохождения нитей, 2 – домен связывания Zn2+, 3 – С-концевой домен
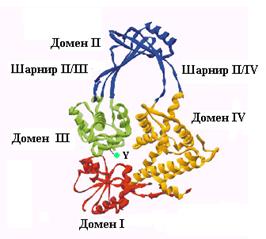
Рис. 2.27. Трехмерная структура фрагмента остатков 32-509 ДНК-топоизомеразы I E. coli.
Указано положение доменов I (остатки 32-63 и 72-157), II (остатки 214-278, 406-433 и 438-475), III (остатки 279-405 и 433-437) и IV (остатки 64-71, 158-213 и 476-560) и междоменных шарниров II/III II/IV, а также каталитического остатка тирозина (Y)
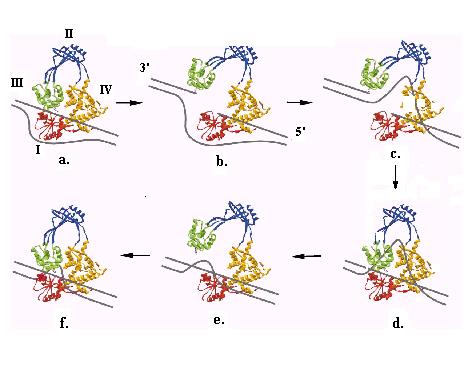
Рис. 2.28. Последовательныке стадии релаксации одного витка негативно суперспирализованной ДНК ДНК-топозомеразой I E. coli.
I, II, III и IV – домены топоизомеразы I, указанные на рис. 2.27. Отмечено положение 3’- и 5’-концов разрезаемой нити ДНК
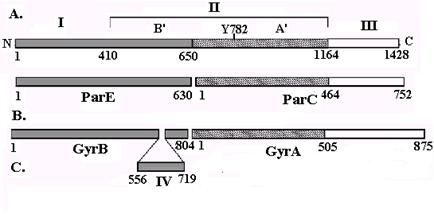
Рис. 2.29. Организация доменов ДНК-топоизомераз типа II.
А – ДНК-топоизомераза II S. cerevisiae, B – ДНК-топоизомераза IV E. coli, С – ДНК-гираза E. coli.
I – АТФазный домен, II – домен связывания и расщепления ДНК, состоящий из субдоменов A’ и B’, которые соответствуют N-концевой области GyrA иС-концевой области GyrB ДНК-гиразы E. coli, IV – уникальная вставка в С-концевой области субъединицы GyrB ДНК-гиразы E. coli
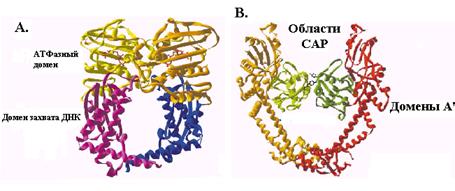
Рис. 2.30. Трехмерная структура субъединиц ДНК-гиразы E. coli.
А – димер N-концевой половины (остатки 1-392) субъединицы GyrB,
В – димер фрагмента остатков 30-522 субъединицы GyrA
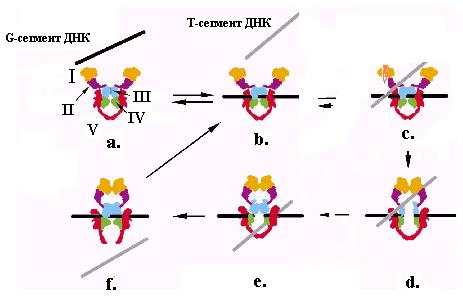
Рис. 2.31. Механизм переноса Т-сегмента ДНК через расщепляемый G-сегмент ДНК-топоизомеразами типа IIA.
I – АТФазные домены (N-ворота), II – домены захвата ДНК, III – области B’, IV -
области САР, V – C-ворота. G-сегмент ДНК изображен черной прямой, а Т-фрагмент ДНК – серой прямой. N-ворота открыты на стадиях а-с и закрыты на стадиях d-f, а С-ворота открыты только на стадии f
— Регулярная проверка качества ссылок по более чем 100 показателям и ежедневный пересчет показателей качества проекта.
— Все известные форматы ссылок: арендные ссылки, вечные ссылки, публикации (упоминания, мнения, отзывы, статьи, пресс-релизы).
— SeoHammer покажет, где рост или падение, а также запросы, на которые нужно обратить внимание.
SeoHammer еще предоставляет технологию Буст, она ускоряет продвижение в десятки раз, а первые результаты появляются уже в течение первых 7 дней. Зарегистрироваться и Начать продвижение
ЛИТЕРАТУРА
1. Matson S.W. DNA helicases of Escherichia coli // Progr. Nucleic Acids Res. Mol. Biol., v. 40, 289-326, 1991
2. Lohman T.M. Helicase-catalyzed DNA unwinding // J. Biol. Chem., v. 268, 2269-2272, 1993
3. West S.C. DNA helicases: new breeds of translocating motors and molecular pumps // Cell, v. 86, 177-180, 1996
4. Lohman T.M., Bjornson K.P. Mechanism of helicase-catalyzed DNA unwinding // Ann. Rev. Biochem., v. 65, 169-214, 1996
5. Patel S.S., Picha K.M. Structure and function of hexameric helicases // Ann. Rev. Biochem., v. 69, 651-697, 2000
6. Hall M.C., Matson S.W. Helicase motifs: the engine that powers DNA unwinding // Mol. Microbiol., v. 34, 867-877, 1999
7. Kersey S.E., Labib K. MCM proteins: evolution, properties, and role in DNA replication // Biochim. Biophys. Acta, v. 1398, 113-136, 1998
8. Tye B.K., Sawyers S. The hexameric eukaryotic MCM helicase: building symmetry from nonidentical parts // J. Biol. Chem., v. 275, 34833-34836, 2000
9. Labib K., Diffley J.F.X. Is the MCM2-7 complex the eukaryotic DNA replication fork helicase? // Curr. Opin. Genet/ Devel., v. 10, 64-70, 2001
10. von Hippel P.H., Delagoutte E. A general model for nucleic acids helicase and their “coupling” within macromolecular machines // Cell, v. 104, 177-190, 2001
11. Chase J.W., Williams K.R. Single-stranded DNA binding proteins required for DNA replication // Ann. Rev. Biochem., v. 55, 103-136, 1986.
12. Lohman T.M, Bujalowski W., Overman L.B. E. coli single strand binding protein: a new look at helix-stabilizing proteins // Trends Biochem. Sci., v. 13, 250-255, 1988.
13. Wold M.S. Replication protein A: a heterodimeric, single stranded DNA-binding protein required for eucaryotic DNA metabolism // Ann. Rev. Biochem., v. 66, 61-92, 1997.
14. Raghunathan S., Ricard C.S., Lohman T.M., Waksman G. Crystal structure of the homo-tetrameric DNA binding domain of Escherichia coli single-stranded DMA binding protein determined by multiwavelength x-ray diffraction on the selenomethionine protein at 2.9-A resolution // Proc. Nat. Acad. Sci. USA, v. 94, 6652-6657, 1997.
15. Shamoo Y., Friedman A.M., Parsons M.R., Konigsberg W.H., Steitz T.A. Crystal structure of a replication fork single-stranded DNA binding protein (T4 gp32) complexed to DNA // Nature, v. 376, 362-366, 1995.
— Разгрузит мастера, специалиста или компанию;
— Позволит гибко управлять расписанием и загрузкой;
— Разошлет оповещения о новых услугах или акциях;
— Позволит принять оплату на карту/кошелек/счет;
— Позволит записываться на групповые и персональные посещения;
— Поможет получить от клиента отзывы о визите к вам;
— Включает в себя сервис чаевых.
Для новых пользователей первый месяц бесплатно. Зарегистрироваться в сервисе
16. Bochkareva E., Belegu V., Korolev S., Bochkarev A. Structure of the major single stranded DNA-binding domain of replication protein A suggests a dynamic mechanism for DNA binding // EMBO Journal, v. 20, 612-618, 2001.
17. Griep M.A. Primase structure and function // Indian J. Biochem. Biophys., v. 32, 171-178, 1995.
18. Arezi B., Kuchta R.D. Eucaryotic DNA primase// Trens Biochem. Sci., v. 25, 572-576, 2000.
19. Frick D.N., Richardson C.C. DNA primases // Ann. Rev. Biochem., v. 70, 39-80, 2001.
20. Aravind L., Leipe D.D., Koonin E.V. Toprim – a conservative domain in type IA and II topoisomerases, DnaG-type primases, OLD family nucleases and RecR proteins // Nucl. Acids Res., v. 26, 4205-4213, 1998.
21. Lehman I.R. DNA ligase: structure, mechanism, and function // Science, v. 186, 790-797, 1974
22. Lindahl T., Barnes D.E. Mammalian DNA ligases // Ann. Rev. Biochem., v. 61, 285-281, 1992.
23. Doherty A.J., Suh S.W. Structural and mechanistic conservation in DNA ligases // Nucleic Acids Res., v. 28, 4051-4058, 2000
24. Shuman S., Schwer B. RNA capping enzyme and DNA ligase: modular architecture and functional implications // Mol. Microbiol., v. 17, 405-410, 1995.
25. Lee J.Y., Chang C., Song H.K. et al. Crystal structure of NAD+-dependent DNA ligase: modular architecture and functional implications // EMBO Journal, v. 19, 1119-1129, 2000.
26. Postow L., Crisona N.J., Peter B.J., Hardy C.D., Cozzarelli N.R. Topological challenges to DNA replication // Proc. Nat. Acad. Sci. USA, v. 98, 8219-8236, 2001.
27. Wang J.C. DNA topoisomerases // Ann. Rev. Biochem., v. 65, 635-692, 1996
28. Berger J.M. Structure of DNA topoisomerases // Biochim. Biophys. Acta, v. 1400, 3-18, 1998
29. Champoux J.J. DNA topoisomerases: structure, function, and mechanism // Ann. Rev. Biochem., v. 70, 369-413, 2001.
30. Tse-Dinh Y.-C. Bacterial and archeal type I topoisomerases // Biochim. Biophys. Acta, v. 1400, 19-27, 1998.
31. Levine C., Hiasa H., Marians K.J. DNA gyrase and topoisomerase IV: biochemical activities, physiological roles during chromosome repkication, and drug sensitivities // Biochim. Biophys. Acta, v. 1400, 29-43, 1998.
32. Nitiss J.L. Investigating the biological functions of DNA topoisomerases in eukariotic cells // Biochim. Biophys. Acta, v. 1400, 63-81, 1998.
ГЛАВА 3. Инициация репликации хромосомной ДНК
3.1. Инициация репликации хромосомы E. coli
|
|
