
Главная страница Случайная страница
Разделы сайта
АвтомобилиАстрономияБиологияГеографияДом и садДругие языкиДругоеИнформатикаИсторияКультураЛитератураЛогикаМатематикаМедицинаМеталлургияМеханикаОбразованиеОхрана трудаПедагогикаПолитикаПравоПсихологияРелигияРиторикаСоциологияСпортСтроительствоТехнологияТуризмФизикаФилософияФинансыХимияЧерчениеЭкологияЭкономикаЭлектроника
Репликативная вилка E. coli и бактериофага T4
|
|
Во время редупликации ДНК ее дочерние синтезирующиеся цепи расходятся из точки репликации, образуя Y-подобную структуру, называемую репликативной вилкой. Именно в окрестностях этой точки разветвления и локализован функционирующий репликативный комплекс. Современные представления о строении репликативной вилки E. coli схематически изображены на рис. I.46, а. В соответствии с этой моделью, ДНК-хеликаза перемещается в репликативной вилке впереди ДНК-синтезирующего белкового комплекса и расплетает цепи родительской ДНК, причем SSB-белок связывается с образующимися одноцепочечными участками, облегчая процесс расплетения.
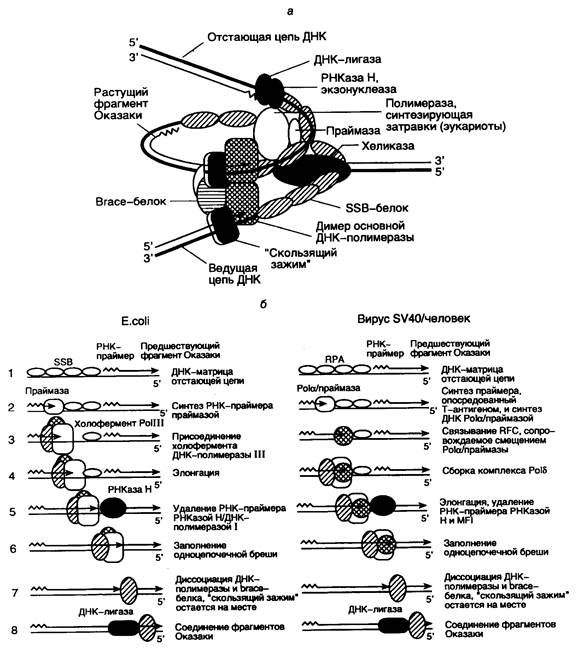
Рис. I.46. Схема функционирующей репликативной вилки E. coli и эукариот и основных этапов репликации ДНК
а – репликативная вилка с основными белками репликации, в которой димер ДНК-полимеразы синхронно реплицирует обе цепи ДНК; б – этапы репликации (1–8) отстающей цепи ДНК. Этап 7 у эукариот является гипотетическим
Из-за антипараллельности и комплементарности цепей ДНК механизм репликации этих двух цепей существенно различается. Действительно, ДНК-полимераза обладает способностью синтезировать ДНК только в одном направлении: от 5’-конца к 3’-концу, перемещаясь вдоль ДНК-матрицы в направлении 3’®5’. В то же время комплементарные цепи ДНК противоположно направлены (антипараллельны), и в силу своих свойств ДНК-полимераза не может реплицировать молекулу ДНК, просто перемещаясь от одного конца матричного дуплекса к другому. Для разрешения этого противоречия репликативный комплекс использует изящный механизм. На одной цепи ДНК синтез новой цепи происходит непрерывно, и образующаяся цепь называется ведущей, тогда как синтез другой цепи осуществляется прерывисто в виде коротких фрагментов, получивших название фрагментов Оказаки в честь ученого, впервые их открывшего. Эта вновь синтезируемая цепь ДНК называется отстающей. И хотя фрагменты Оказаки также синтезируются в направлении 5’®3’, перемещение работающей ДНК-полимеразы вдоль матричной цепи ДНК при синтезе каждого индивидуального фрагмента Оказаки должно быть противоположным тому, которое имеет место в случае синтеза ведущей цепи. Образующиеся фрагменты Оказаки отстающей цепи далее соединяются друг с другом с помощью ДНК-лигазы.
— Регулярная проверка качества ссылок по более чем 100 показателям и ежедневный пересчет показателей качества проекта.
— Все известные форматы ссылок: арендные ссылки, вечные ссылки, публикации (упоминания, мнения, отзывы, статьи, пресс-релизы).
— SeoHammer покажет, где рост или падение, а также запросы, на которые нужно обратить внимание.
SeoHammer еще предоставляет технологию Буст, она ускоряет продвижение в десятки раз, а первые результаты появляются уже в течение первых 7 дней. Зарегистрироваться и Начать продвижение
Репликативный комплекс, который осуществляет синтез ведущей цепи ДНК, включает в себя минимальный (кор-) фермент ДНК-полимеразы III (белок 43 в случае фага Т4), подвижный связывающий b-белок с молекулярной массой 41 кДа (" sliding clamp", белок 45 у фага Т4) и белки g-комплекса, состоящего из пяти полипептидов gdd‘cy (скрепляющие белки – brace proteins). Функциональным аналогом белков g-комплекса у бактериофага Т4 служит комплекс белковых продуктов генов 44/62. При облучении клеток E. coli УФ-светом в них индуцируется синтез укороченного b*-белка (26 кДа), который является продуктом того же гена, что и b-субъединица холофермента ДНК-полимеразы III. По-видимому, функциональная роль b*-белка заключается в обеспечении репликации ДНК на матрице, поврежденной УФ-светом.
При синтезе ведущей цепи ДНК репликативный комплекс E. coli функционирует весьма эффективно с высокой процессивностью. Напомним, что мерой процессивности является длина фрагмента вновь синтезированной макромолекулы, которую комплекс (или индивидуальные ферменты) способен образовывать в одном цикле, не диссоциируя от матрицы. Установлено, что холофермент ДНК-полимеразы III, в состав которого входят минимальный фермент (субъединицы a, q и e), b-белок, белки g-комплекса и t-белок, синтезирует ведущую цепь ДНК длиной в 50000 нуклеотидов со скоростью > 500 нуклеотидов в секунду в одном цикле, ни разу не диссоциируя от ДНК-матрицы. Точность репликации ДНК холоферментом ДНК-полимеразы III поражает воображение. Частота ошибочных включений нуклеотидов не превышает 10-9–10-10 на нуклеотид за один раунд репликации. В то же время очищенные каталитические субъединицы реплицируют ДНК с пониженной точностью. В частности, изолированная a-субъединица допускает ошибки в опытах in vitro с частотой ~610-1 на нуклеотид за один раунд репликации. Частота ошибок, возникающих в ДНК, облученной УФ-светом, одна и та же в ведущей и отстающей цепях вновь синтезируемой ДНК in vivo.
— Разгрузит мастера, специалиста или компанию;
— Позволит гибко управлять расписанием и загрузкой;
— Разошлет оповещения о новых услугах или акциях;
— Позволит принять оплату на карту/кошелек/счет;
— Позволит записываться на групповые и персональные посещения;
— Поможет получить от клиента отзывы о визите к вам;
— Включает в себя сервис чаевых.
Для новых пользователей первый месяц бесплатно. Зарегистрироваться в сервисе
Роль g-комплекса заключается в распознавании РНК-затравок (праймеров) на матричной ДНК. g-Комплекс связывается с единственным праймером ведущей цепи ДНК или с каждым из праймеров фрагментов Оказаки отстающей цепи, что, в свою очередь, делает возможным присоединение к промаркированным таким образом праймерам минимального фермента ДНК-полимеразы и b-белка (см. рис. I.46, б).
Две молекулы b-белка входят в состав репликативного комплекса вслед за белками g-комплекса, связываясь с ДНК позади белков g-комплекса и оставляя 3’-конец праймера доступным для ДНК-полимеразы. Димер b-белка образует кольцо вокруг молекулы ДНК и стимулирует АТРазную активность белков g-комплекса. Как уже упоминалось выше, функциональный аналог b-белка – продукт гена 45 бактериофага Т4, образует такую же пространственную структуру, охватывающую молекулу ДНК тремя молекулами. Молекулярная масса белка 45 составляет 2/3 от таковой b-мономера, и их аминокислотные последовательности негомологичны друг другу. Тем не менее, четвертичные структуры этих полипептидов и механизмы их функционирования обладают большим сходством.
b-Белки и белки g-комплекса, будучи связанными с дуплексом праймер–матрица, обеспечивают присоединение к этому комплексу минимального фермента ДНК-полимеразы. Затем ДНК-полимераза при наличии доступных четырех дезоксирибонуклеозидтрифосфатов, используя праймер для инициации синтеза ДНК, с высокой эффективностью синтезирует цепь ДНК, комплементарную ДНК-матрице. Те же самые белки участвуют и в синтезе отстающей цепи ДНК. В этом случае прерывистый синтез ДНК многократно инициируется на большом количестве праймеров, и ДНК синтезируется в виде фрагментов Оказаки длиной ~1000 нт. Синтез затравок, представляющих собой короткие последовательности РНК, обеспечивает продукт гена dnaG (белок 61 фага Т4). ДНК-полимераза начинает элонгацию цепей ДНК, присоединяя первый дезоксирибонуклеозидмонофосфат к 3’-концевому нуклеотиду РНК-затравки. В процессе элонгации участвуют b-белок и белки g-комплекса, которые перемещаются вдоль молекулы ДНК вместе с каталитической субъединицей ДНК-полимеразы.
Ведущая и отстающая цепи ДНК реплицируются координированно, что обеспечивается димеризацией ДНК-полимеразных комплексов. В таком димере, который образуется при участии t-белка, один ДНК-полимеразный комплекс осуществляет непрерывный синтез ведущей цепи ДНК, а другой – фрагментов Оказаки отстающей цепи. Для димеризации ДНК-полимеразы III E. coli необходим t-белок, в то время как продукт гена 43 бактериофага Т4, по-видимому, изначально находится в виде димера. Другое различие репликативных комплексов E. coli и фага Т4 заключается в том, что холофермент ДНК-полимеразы E. coli (субъединицы aeq·gdd‘·cy·t·b) сохраняется в виде стабильного комплекса и в отсутствие ДНК, тогда как холофермент Т4-ДНК-полимеразы существует только в присутствии матрицы.
Необычность ситуации во время синтеза ДНК в репликативной вилке заключается в том, что один и тот же белковый комплекс осуществляет как высоко процессивный непрерывный синтез ведущей цепи ДНК, так и прерывистый синтез фрагментов Оказаки отстающей цепи, претерпевая во втором случае периодическую диссоциацию от матрицы для инициации синтеза ДНК с каждого нового праймера. Для объяснения такого парадокса предположили, что холофермент ДНК-полимеразы способен узнавать 5’-конец каждого РНК-праймера, встречающегося после завершения синтеза очередного фрагмента Оказаки во время образования отстающей цепи ДНК в процессе репликации. Недавно с помощью оригинального экспериментального подхода удалось решить этот вопрос. В участок полипептидной цепи b-белка, контактирующий с минимальным ферментом ДНК-полимеразы III, методами генной инженерии ввели аминокислотную последовательность, узнаваемую и фосфорилируемую протеинкиназой. Измеряя скорость фосфорилирования этих сайтов в условиях избытка протеинкиназы во время синтеза ДНК in vitro, определили кинетику ассоциации и диссоциации комплексов b-белок–ДНК-полимераза по изменению уровня защищенности сайтов фосфорилирования от действия протеинкиназы. Полученные результаты интерпретировали таким образом, что во время связанного с синтезом фрагментов Оказаки перемещения минимального фермента ДНК-полимеразы и g-комплекса вдоль одноцепочечной ДНК-матрицы, покрытой SSB-белком, оба белка прочно связаны с b-белком и матрицей. При встрече репликативного белкового комплекса с дуплексом, образованным матричной ДНК и РНК-затравкой, b-белок остается связанным с вновь синтезированной ДНК, а у отделившихся ДНК-полимеразы и белков g-комплекса появляется возможность вступить в новый цикл синтеза фрагмента Оказаки путем взаимодействия с очередным дуплексом РНК-затравка–матрица. При этом вхождение ДНК-полимеразы в новый репликативный комплекс облегчается наличием в нем b-белка и белков g-комплекса, ассоциированных с очередным РНК-праймером. Таким образом, холофермент ДНК-полимеразы III обладает способностью распознавать молекулярное окружение, создаваемое матричной ДНК, осуществлять терминацию синтеза ДНК при наличии сигнала в виде дуплекса ДНК–затравка и реинициировать синтез ДНК на следующем праймере. В итоге одна и та же молекула ДНК-полимеразы III в составе реплицирующего комплекса способна проводить синтез всех фрагментов Оказаки отстающей цепи реплицируемой ДНК, последовательно осуществляя инициацию, терминацию и реинициацию синтеза каждого из них.
После очередной терминации синтеза ДНК отстающей цепи 3’-конец вновь синтезированной ДНК оказывается вплотную приближенным к 5’-концу праймера следующего фрагмента Оказаки. Для соединения двух фрагментов с помощью ДНК-лигазы необходимы предварительное удаление РНК-праймера и достройка цепи ДНК в образующейся бреши. РНК-затравка удаляется с помощью РНКазы H, нуклеазы, специфически расщепляющей РНК в ДНК–РНК-гибридах, и(или) с участием 5’®3’-экзонуклеазы ДНК-полимеразы I. Во втором случае одновременно с удалением праймера происходит застройка образующейся бреши той же ДНК-полимеразой. В итоге два соседних фрагмента Оказаки вплотную приближаются друг к другу и оказываются отделенными лишь одноцепочечным разрывом, который может репарироваться ДНК-лигазой. В настоящее время остается открытым вопрос о механизмах координации удаления РНК-праймеров из фрагментов Оказаки с самим процессом репликации ДНК.
Кроме вышеупомянутых дуплексов праймеры–ДНК, холоферменты бактериальных и фаговых ДНК-полимераз, по-видимому, способны адекватно реагировать на другие стерические препятствия, возникающие на пути следования вдоль реплицируемой молекулы ДНК. В частности, ДНК-полимераза фага Т4 в процессе репликации может расходиться с транскрибирующими ту же ДНК молекулами РНК-полимеразы, не диссоциируя из репликативного комплекса и не вытесняя РНК-полимеразу с матрицы. Кроме того, репликативный комплекс может распознавать повреждения ДНК, возможно, маркированные специфическими белками, и прекращать репликацию соответствующего участка, останавливаясь или диссоциируя от матрицы. Репликация таких участков ДНК возобновляется после ликвидации повреждений ферментами репаративной системы. Для диссоциировавшей ДНК-полимеразы это становится возможным благодаря тому, что 3’-конец вновь синтезированной цепи ДНК в месте остановки репликации остается связанным с b-белком, который облегчает повторное вхождение диссоциировавшей ДНК-полимеразы в репликативный комплекс.
|
|
